COVID-19 Nukleotidsequenz anschauen: Unterschied zwischen den Versionen
| Zeile 80: | Zeile 80: | ||
| − | So, jetzt schauen wir uns ein Feature von Covid genauer an. Ich | + | So, jetzt schauen wir uns ein Feature von Covid genauer an. Ich wähle als Beispiel die Sequenz ORF3a aus. |
[[Datei:snapgene_select_orf3a.png|300px]] | [[Datei:snapgene_select_orf3a.png|300px]] | ||
| Zeile 115: | Zeile 115: | ||
| − | Wenns fertig ist sieht es so aus | + | Wenns fertig ist, sieht es so aus |
[[Datei:phyre2_orf3a_feature_done.png|500px]] | [[Datei:phyre2_orf3a_feature_done.png|500px]] | ||
Version vom 8. März 2020, 19:53 Uhr
Vorwort Erstmal, Ich bin kein Biologe, nur ein neugieriger Bastler ;D Folgende Anleitung basiert aus einen Facebook Video, hochgeladen von einen BioClub Tokyo BioHacker (JiKong Chan). https://www.facebook.com/groups/BioClubPublic/permalink/2909493445774566/
Oberes Workshop erklärt uns folgendes:
- Wie man ein Sequenz mit der Software SnapGene laden und anschauen kann.
- Aus SnapGene Enzym Sequenz exportieren und mit Phyre2 das Protein berechnen.
- Mit Jmol das berechnet Protein in 3D anschauen.
Ok, so jetzt befriedigen wir unsere Neugier.
Als erstes benötigen wir paar Software
Erstmal benötigten wir Software (alles Freie Software)
SnapGene
https://www.snapgene.com/snapgene-viewer/
Mit dieser Software kann man sich Gen Sequenzen anschauen, genauer DNA Sequenz. Basierend der Sequenzen wird auch die Enzyme angezeigt.
Freie Cloud Server Dienst um aus Enzym Sequenz ein Protein zu berechnen. Je nach Komplexität kann es mehrer Stunden dauern
http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index
Software um Moleküle zu visualisieren.
https://sourceforge.net/projects/jmol/
Als letztes brauche wir das complete Genom unser Virus
Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome:
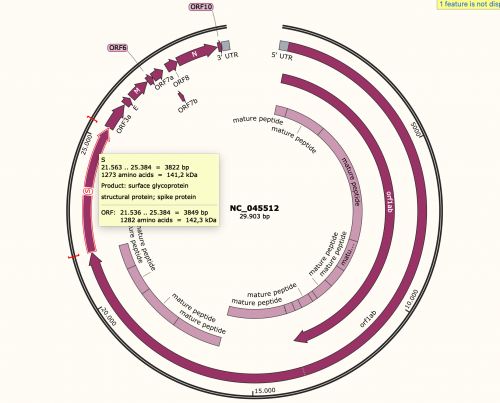
Von hier benötigen wir die "accession" ID. Mit diesen ID kann man sich die Sequenz aus einer Repository später in SnapGene runterladen. Für unser Virus ist es die accession ID -> NC_045512
Step-by-Step Anleitung wie man in SnapGene ein Genom lädt
Wie oben beschrieben besorgen wir uns erstmal die accession ID (NC_045512). Mit der accession ID könne wir es importieren Files -> Import ->NCBI Sequenz .
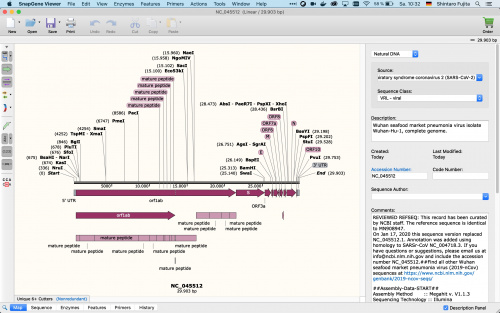
Wenn wir erfolgreich importiert haben müsste es ungefähr so aussehen.
Wir ändern die Ansicht damit es übersichtlicher ist.
Nach abwählen der oben beschriebenen Buttons müsste es so aussehen
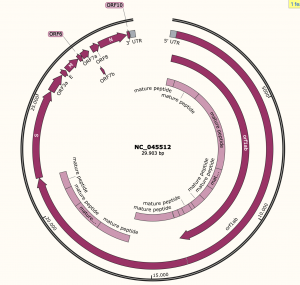
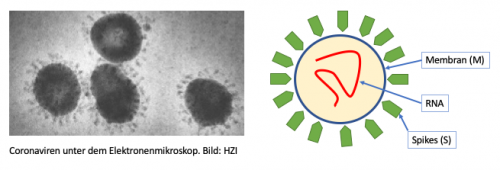
Super vereinfachtes Teardown des Virus.
Was wir mit SnapGene betrachten ist die RNA. Da der RNA quasi eine Bauanleitung des Virus ist, kann man auch die einzelne Bauanleitungen der Komponenten in der RNA finden. In den oberen Bild habe Ich die Kürzel der Komponenten im Klammer gesetzt. Beispiel "S" für den Spike. In SnapGene ist es auch mit S gekenzeichnet. Der Spike ist die Komponente was den Virus befähigt seine RNA ins Wirtszellen einzuschleusen.
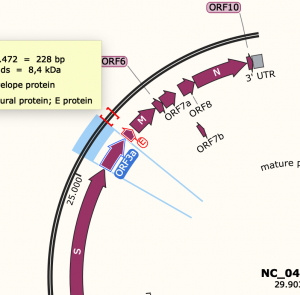
So, jetzt schauen wir uns ein Feature von Covid genauer an. Ich wähle als Beispiel die Sequenz ORF3a aus.
Danach könne wir uns die Sequenz unterschiedlich anschauen. Wir wechseln die Ansicht mit den Buttons unten Link.
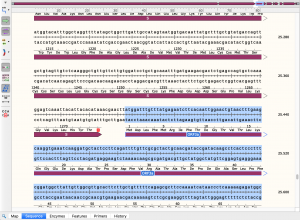
DNA Sequenz Ansicht von ORF3a. Ja eigentlich hat ja ein Virus nur ein RNA, aber diese software ist für DNA gedacht, ist aber nicht schlimm, die RNA ist die obere hälfte.
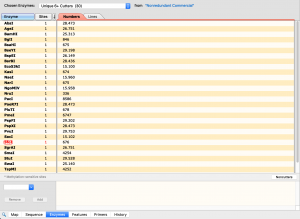
Die Enzyme die durch die ORF3a Sequenz generiert wird.
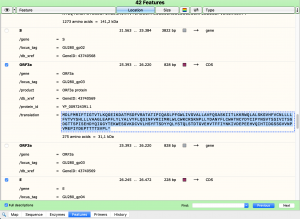
Feauture Informationen von ORF3a. Hier kopieren wir uns die "/translation" Komplet. Soweit Ich es verstanden habe ist es ein art G-Code für Enzyme. Diese brauchen wir für den nächsten Schritt.
Jetzt werden wir anhand der Enzym Sequenz von Covid's ORF3a diese durch ein Cloud Server uns das Endprodukt, ein Protein, berechnen lassen. Dazu öffnen wir folgende Seite.
http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index
Dort geben wir die Sequenz, und eine E-Mail Adresse an. Die E-Mail Adresse wird benötigt, da die Berechnung je nach Komplexität lange dauert. Bei ORF3a wurde nur ca. über eine Stunde geschätzt, da diese ein recht einfaches Protein ist. Beim Spike kann es über 8 Stunden dauern.
Wenns fertig ist, sieht es so aus