Vorwort Erstmal, Ich bin kein Biologe, nur ein neugieriger Bastler ;D Folgende Anleitung basiert aus einen Facebook Video, hochgeladen von einen BioClub Tokyo BioHacker. https://www.facebook.com/groups/BioClubPublic/permalink/2909493445774566/
Oberes Workshop erklärt uns folgendes:
- Wie man ein Sequenz mit der Software SnapGene laden und anschauen kann.
- Aus SnapGene Enzym Sequenz exportieren und mit Phyre2 das Protein berechnen.
- Mit Jmol das berechnet Protein in 3D anschauen.
Ok, so jetzt befriedigen wir unsere Neugier. Als erstes benötigen wir paar Software
Erstmal benötigten wir Software (alles Freie Software)
SnapGene https://www.snapgene.com/snapgene-viewer/ Mit dieser Software kann man sich Gen Sequenzen anschauen, genauer DNA Sequenz. Basierend der Sequenzen wird auch die Enzyme angezeigt.
Freie Cloud Server Dienst um aus Enzym Sequenz ein Protein zu berechnen. Je nach Komplexität kann es mehrer Stunden dauern http://www.sbg.bio.ic.ac.uk/phyre2/html/page.cgi?id=index
Software um Moleküle zu visualisieren.
https://sourceforge.net/projects/jmol/
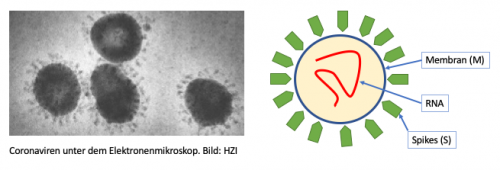
Als letztes brauche wir das complete Genom unser Virus Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome: https://www.ncbi.nlm.nih.gov/nuccore/NC_045512?fbclid=IwAR0QIfHRx9ktGU6VojsdWvWtRitUCYQSCtD7xsWtqGusS55xVDzw0uigWLs Von hier benötigen wir die "accession" ID. Mit diesen ID kann man sich die Sequenz aus einer Repository später in SnapGene runterladen. Für unser Virus ist es die accession ID -> NC_045512
Step-by-Step Anleitung wie man in SnapGene ein Genom lädt
Wie oben beschrieben besorgen wir uns erstmal die accession ID (NC_045512). Mit der accession ID könne wir es importieren Files -> Import ->NCBI Sequenz .
Wenn wir erfolgreich importiert haben müsste es ungefähr so aussehen.
Wir ändern die Ansicht damit es übersichtlicher ist.